# A tibble: 32 × 11
mpg cyl disp hp drat wt qsec vs am gear carb
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2
6 18.1 6 225 105 2.76 3.46 20.2 1 0 3 1
7 14.3 8 360 245 3.21 3.57 15.8 0 0 3 4
8 24.4 4 147. 62 3.69 3.19 20 1 0 4 2
9 22.8 4 141. 95 3.92 3.15 22.9 1 0 4 2
10 19.2 6 168. 123 3.92 3.44 18.3 1 0 4 4
# ℹ 22 more rowsR语言如何绘制相关性图
前言
本篇是R语言ggplot2系列包手搓带聚类,条带的半相关性图。
什么是相关性图?
相关性是指两个或多个变量之间的关系或相互影响程度。若两组的值一起增大,我们称之为正相关,若一组的值增大时,另一组的值减小,我们称之为负相关。其值介于-1与1之间,即越接近1,越正相关;越接近-1,越负相关。
常见的有两种计算相关性的算法:皮尔逊相关性(pearson)和斯皮尔曼相关性(spearman)。皮尔逊相关性最常用,适合正态分布的数据。斯皮尔曼相关性是秩相关,不受极大极小值的影响。
在组学中的应用,例如,两个技术性重复实验的结果相关性很低,则说明数据有异常。
需要注意的地方,做样本之间的相关性的时候,蛋白之间要对应,不可随意打乱顺序。数据中有缺失值的情况,通常用”pairwise.complete.obs”算法处理缺失值,即两两配对删除缺失值。
相关性图就是,用图的形式展现出相关性的结果。
绘图前的数据准备
这里就用R语言自带的示例数据了,mtcars
R语言如何绘制相关性图
# 代码来源:https://www.r2omics.cn/
# 加载必需R包(若未安装,需先运行:install.packages(c("ggplot2", "dplyr", "scales", "aplot", "ggtree", "psych", "tibble", "tidyr", "openxlsx")))
library(ggplot2) # 核心绘图包,用于绘制热图、分组标签等
library(dplyr) # 数据处理包,用于筛选、排序、聚合等操作
library(scales) # 颜色缩放工具包(本代码未直接调用,但为相关性热图常用依赖)
library(aplot) # 图形组合包,实现多图拼接(上下/左右插入)
library(ggtree) # 聚类树绘制包,用于可视化层次聚类结果
library(psych) # 相关性分析包,提供corr.test函数计算相关系数和p值
library(tibble) # 数据框处理包,提供rownames_to_column等行名转列功能
library(tidyr) # 数据格式转换包,提供pivot_longer实现宽格式转长格式
library(openxlsx) # 文件保存包,用于将相关性矩阵导出为Excel
library(ggstyle) # 提供更多主题和配色方案devtools::install_github("sz-zyp/ggstyle")
# ===================== 1. 模拟数据(可替换为真实数据)=====================
set.seed(123) # 固定随机种子,确保每次运行生成相同模拟数据(结果可重复)
n <- 50 # 样本数量(数据框行数:50个样本)
var_num <- 10 # 变量数量(数据框列数:10个变量)
# 生成50行×10列的正态分布数据(均值0,标准差1)
df = mtcars
# 定义变量分组信息(10个变量分为2组:A组5个,B组5个)
dfGroup <- data.frame(
"Sample" = colnames(df), # 变量名(与df的列名一致,此处命名为Sample仅为方便后续绘图)
"Group" = c(rep("A", 5), rep("B", 6)) # 分组:前5个变量为A组,后5个为B组
)
# ===================== 2. 计算变量间相关性矩阵 =====================
ct <- psych::corr.test(
x = df, # 输入数据(行=样本,列=变量)
use = "pairwise", # 缺失值处理方式:成对删除(仅删除存在缺失值的成对观测)
method = "pearson", # 相关性计算方法:皮尔逊相关(适用于正态分布数据)
adjust = "BH", # p值矫正方法:Benjamini-Hochberg(控制假发现率)
ci = FALSE # 不计算相关系数的置信区间(简化输出)
)
# ct的核心输出(按需查看):
# ct$r:相关性系数矩阵(10×10,变量间两两相关系数)
# ct$p:原始p值矩阵(未矫正的显著性检验结果)
# ct$p.adj:矫正后的p值矩阵(BH矫正后,更稳健)
# ct$n:每个相关系数的有效样本量
# 提取相关性系数矩阵,转换为数据框格式
cor_matrix <- data.frame(ct$r)
# ===================== 3. 变量层次聚类(用于热图排序)=====================
# 计算变量间距离矩阵:1-相关系数(相关系数越近,距离越小)
row_dist <- dist(1 - cor_matrix)
# 层次聚类(默认方法:ward.D2,最小化组内方差)
row_clust <- hclust(row_dist)
# 绘制垂直聚类树(用于热图左侧显示)
pTreeY <- row_clust %>%
ggtree::ggtree(
layout = "rectangular", # 矩形布局(清晰展示聚类结构)
branch.length = "none" # 不显示分支长度(仅关注聚类顺序)
)
# + geom_tiplab(size=2) # 可选:添加聚类树叶节点标签(变量名)
# 提取聚类后的变量顺序(关键:用于后续热图的行列排序)
myOrder <- pTreeY$data %>%
arrange(angle) %>% # 按角度排序(ggtree内部排序逻辑,确保叶节点顺序正确)
filter(isTip) %>% # 筛选叶节点(仅保留变量对应的节点)
pull(label) # 提取叶节点标签(变量名),得到聚类后的顺序
# 绘制水平聚类树(用于热图顶部显示)
pTreeX <- pTreeY +
ggtree::layout_dendrogram() # 转换为水平布局(适配热图顶部位置)
# + geom_tiplab(size=2) # 可选:添加聚类树叶节点标签(变量名)
# ===================== 4. 相关性矩阵预处理(排序+上三角提取)=====================
# 按聚类结果重新排序相关性矩阵(行:逆序myOrder,列:myOrder,确保与聚类树匹配)
cor_matrix_ordered <- cor_matrix[rev(myOrder), myOrder]
# 生成上三角相关性矩阵(下三角设为NA,绘图时不显示,避免重复信息)
cor_matrix_upper <- cor_matrix[myOrder, myOrder] # 按聚类顺序排序原始相关矩阵
# lower.tri:标记下三角元素(diag=FALSE:不包含对角线),设为NA
cor_matrix_upper[lower.tri(cor_matrix_upper, diag = FALSE)] <- NA
# 转换为ggplot2所需的长格式数据(热图绘图要求)
cor_long <- cor_matrix_upper %>%
tibble::rownames_to_column("Var1") %>% # 行名(变量1)转为列Var1
pivot_longer(
-1, # 除Var1外的所有列(即变量2)
names_to = "Var2", # 列名转为Var2(变量2)
values_to = "Correlation", # 数值转为Correlation(相关系数)
values_drop_na = TRUE # 删除NA值(下三角部分,不绘图)
)
# 确保Var1和Var2为因子类型,并固定 levels 为聚类顺序(避免绘图时自动排序)
cor_long$Var1 <- factor(cor_long$Var1, levels = colnames(cor_matrix_ordered))
cor_long$Var2 <- factor(cor_long$Var2, levels = colnames(cor_matrix_ordered))
# 验证排序是否正确(可选)
# cor_long$Var1 %>% levels()
# cor_long$Var2 %>% levels()
# ===================== 5. 绘制核心图形组件 =====================
# 注意:需提前定义两个全局变量(用户代码遗漏,此处补充说明)
borderSize <- 0.2 # 热图单元格边框宽度(可自定义,如0.1~0.5)
myTextSize <- 8 # 坐标轴文字大小(可自定义,如6~10)
# 5.1 上三角相关性热图(核心图p1)
p1 <- ggplot(cor_long, aes(x = Var1, y = Var2, fill = Correlation)) +
geom_tile(
color = "white", # 单元格边框颜色(白色,分隔单元格)
size = borderSize, # 边框宽度(提前定义的borderSize)
na.rm = TRUE # 忽略NA值(不绘制下三角)
) +
# 双向颜色映射(适配相关系数[-1,1])
scale_fill_gradient2(
low = "#0001c3", # 负相关颜色(深蓝色)
mid = "white", # 无相关颜色(白色,midpoint=0)
high = "#cb0403", # 正相关颜色(深红色)
midpoint = 0, # 颜色中点(相关系数=0)
limit = c(-1, 1), # 颜色范围(覆盖所有相关系数)
space = "Lab", # 颜色空间(Lab空间颜色过渡更均匀)
name = "Cor" # 图例名称(相关系数)
) +
theme_void() + # 清除默认主题(无坐标轴、网格线等)
scale_y_discrete(position = "left") + # y轴标签放在左侧(与聚类树对齐)
theme(
axis.ticks = element_blank(), # 隐藏坐标轴刻度线
axis.title = element_blank(), # 隐藏坐标轴标题
panel.grid = element_blank(), # 隐藏网格线
axis.text.y = element_text( # 调整y轴文字(变量名)
size = myTextSize, # 文字大小(提前定义的myTextSize)
hjust = 1 # 文字右对齐(贴近热图)
)
)
# 5.2 顶部分组标签图(p2:显示变量分组A/B)
p2 <- ggplot(dfGroup) +
geom_tile(
aes(
x = factor(Sample, levels = rev(colnames(cor_matrix_ordered))), # x轴:变量(聚类逆序,与顶部聚类树匹配)
y = 1, # y轴固定为1(仅作为色块高度)
fill = Group # 按分组填充颜色
)
) +
theme_void() + # 清除默认主题
theme(
axis.ticks = element_blank(), # 隐藏刻度线
axis.title = element_blank(), # 隐藏标题
panel.grid = element_blank(), # 隐藏网格线
axis.text.x = element_text( # 调整x轴文字(变量名)
angle = 90, # 文字旋转90度(避免重叠)
hjust = 0, # 水平对齐(左对齐)
vjust = 0.5, # 垂直居中
size = myTextSize # 文字大小
)
)+
ggstyle::scale_fill_sci(palette = "Set2",modeColor = "1")
# 5.3 左侧分组标签图(p3:显示变量分组A/B)
p3 <- ggplot(dfGroup) +
geom_tile(
aes(
x = 1, # x轴固定为1(仅作为色块宽度)
y = factor(Sample, levels = colnames(cor_matrix_ordered)), # y轴:变量(聚类顺序,与左侧聚类树匹配)
fill = Group # 按分组填充颜色
)
)+
ggstyle::scale_fill_sci(palette = "Set2",modeColor = "1")
# 定义简化主题函数(统一清除冗余元素)
mythemeF <- function(p) {
p + theme_void() # 应用无坐标轴主题
}
# ===================== 6. 组合图形(两种组合方案)=====================
# 方案1:热图 + 顶部分组标签 + 左侧分组标签(无聚类树)
p <- mythemeF(p1) %>% # 给p1应用简化主题
aplot::insert_top((mythemeF(p2)), height = 0.05) %>% # 顶部插入p2,高度占比5%(避免标签过高)
aplot::insert_left(mythemeF(p3), width = 0.05) # 左侧插入p3,宽度占比5%(避免标签过宽)
p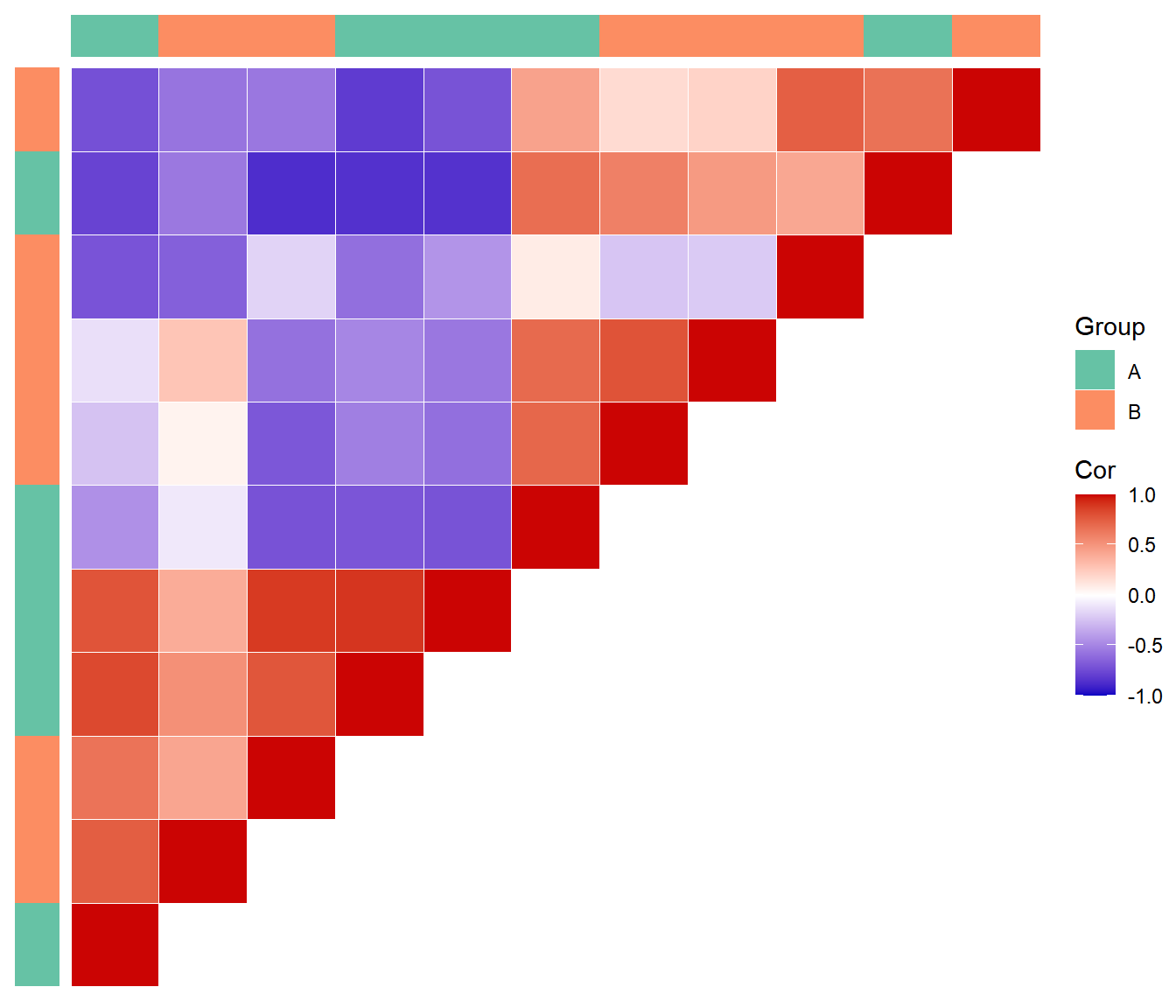
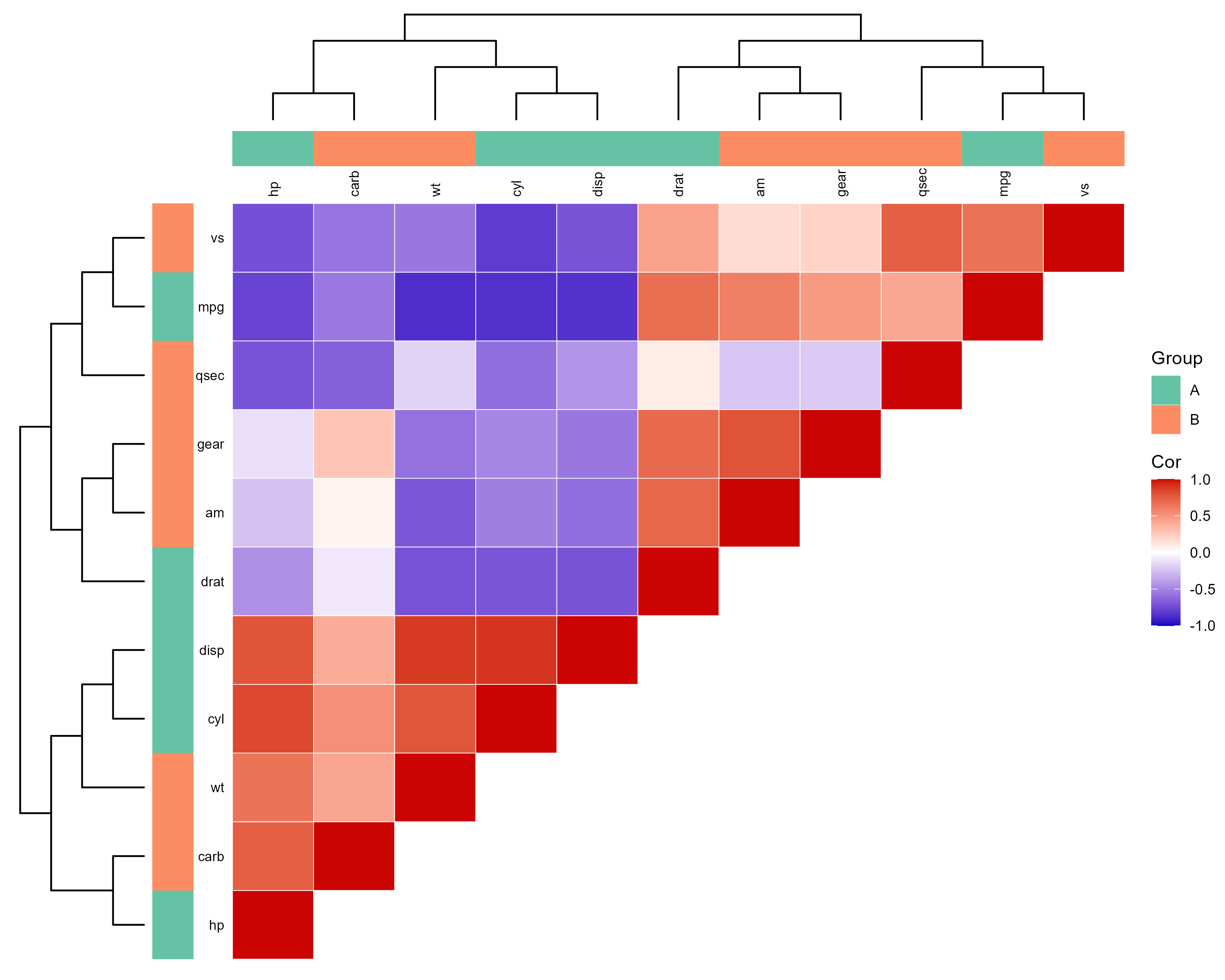
# 方案2:完整组合(热图 + 聚类树 + 分组标签)
pAll <- (p1) %>%
aplot::insert_top((p2), height = 0.05) %>% # 顶部:分组标签(5%高度)
aplot::insert_left(mythemeF(p3), width = 0.05) %>% # 左侧:分组标签(5%宽度)
aplot::insert_top(pTreeX, height = 0.15) %>% # 顶部上方:水平聚类树(15%高度)
aplot::insert_left(pTreeY, width = 0.15) # 左侧左方:垂直聚类树(15%宽度)
pAll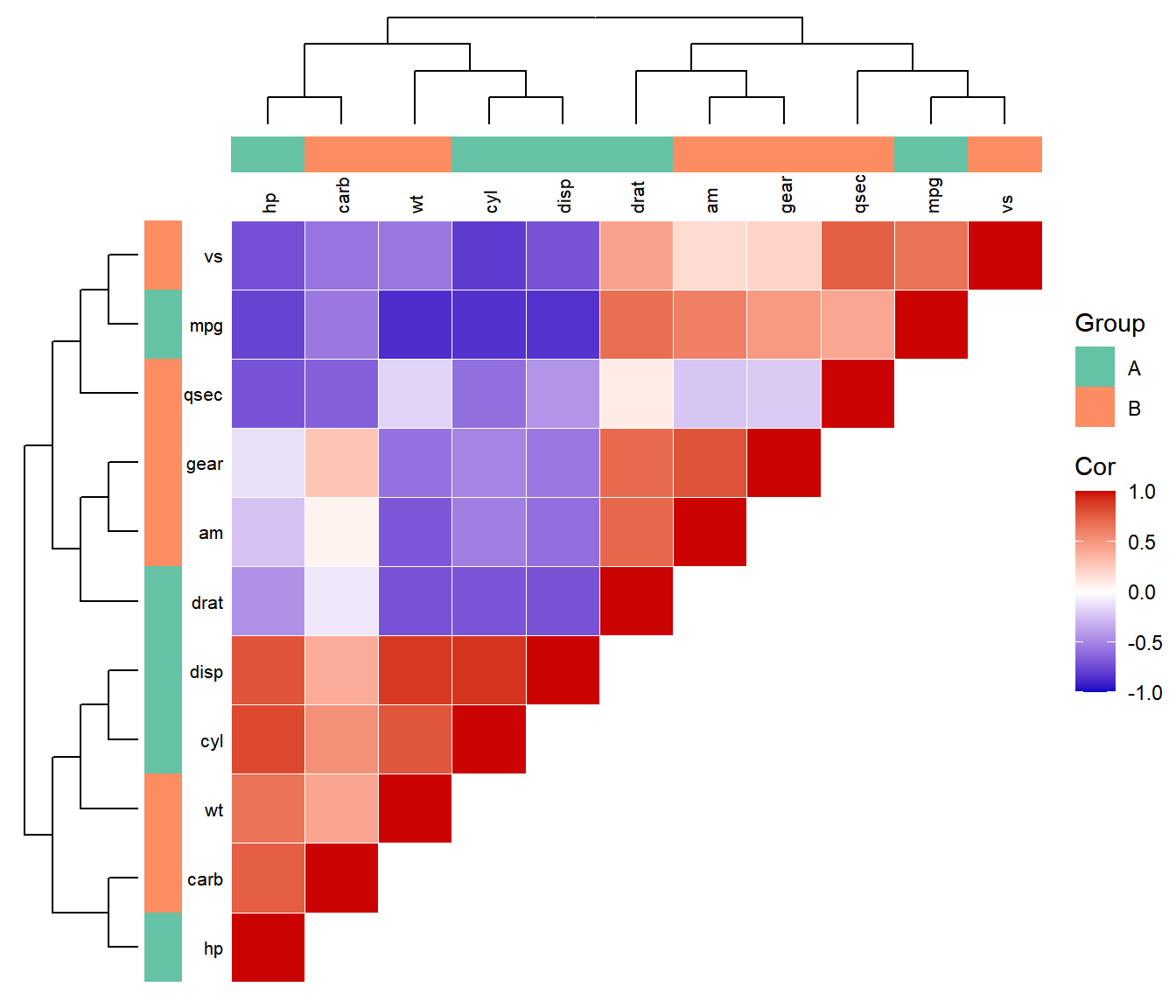
# ===================== 7. 保存结果(文件输出)=====================
# 注意:需提前定义输出路径outPath1(示例:outPath1 <- "你的输出文件夹路径/")
# 保存排序后的相关性矩阵为Excel文件(含行名)
# openxlsx::write.xlsx(
# x = data.frame(cor_matrix_ordered, check.names = FALSE), # 保留原始列名(不自动修改特殊字符)
# file = file.path("sampleCor.xlsx"), # 输出文件路径+名称
# rowNames = TRUE # 保留行名(变量名)
# )
# 保存方案1图形(无聚类树)
# ggsave(
# filename = file.path("样本相关性图.png"), # 图片路径+名称
# plot = p, # 待保存图形
# width = 10, # 图片宽度(英寸)
# height = 8, # 图片高度(英寸)
# dpi = 300 # 分辨率(300dpi,高清)
# )
#
# # 保存方案2图形(带聚类树和分组标签)
# ggsave(
# filename = file.path("样本相关性图_带聚类树和名字.png"), # 图片路径+名称
# plot = pAll, # 待保存图形
# width = 10, # 图片宽度
# height = 8, # 图片高度
# dpi = 300 # 分辨率
# )